5.5 数据插补 EMP_impute
数据插补是指当数据集中存在缺失值时,通过算法对这些缺失值进行估计或填充的过程。其目的是在缺失值的位置上提供合理的估计值,从而在后续的数据分析或建模过程中更好地利用这些数据。EMP_impute 模块基于链式随机森林(Chained Random Forests, CRF)算法进行数据插补,该算法比传统的均值或众数插补方法更具科学性,不仅可以用于插补连续型变量,还可以用于插补分类变量。模块EMP_impute 支持对 assay、rowdata 和 coldata 进行数据插补。
5.5.1 插补样本相关数据coldata
组学项目的coldata是指样本相关数据,经常会出现不同程度数据缺失。导致缺失值的常见原因包括:数据采集过程出现错误或遗漏、受试者中途退出研究、技术问题(例如:设备故障或数据传输错误)等。模块EMP_impute可以基于CRF算法对coldata的缺失值进行插补。
🏷️示例:
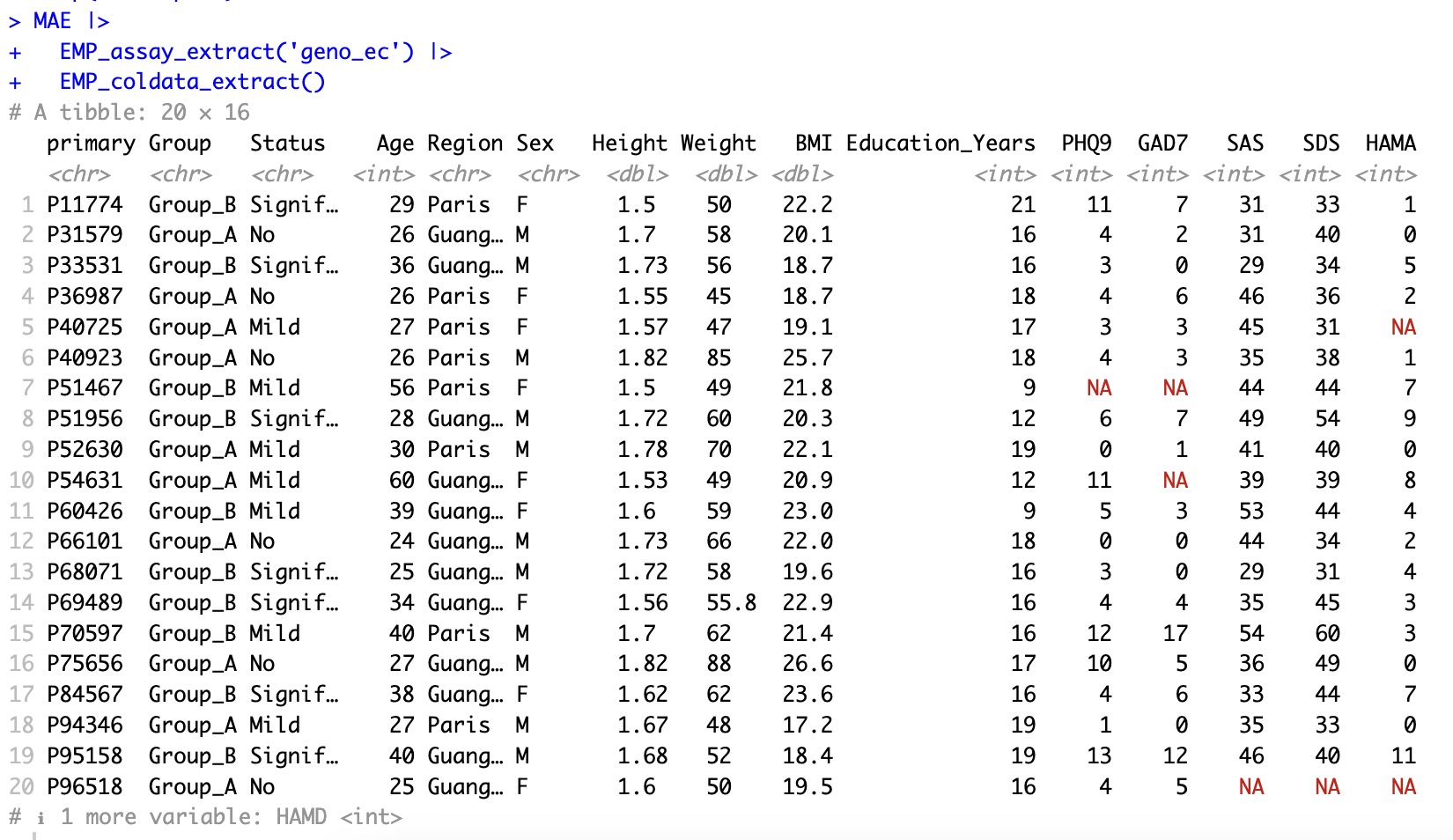
插补前:coldata存在大量缺失值。
MAE |>
EMP_assay_extract('geno_ec') |>
EMP_coldata_extract()
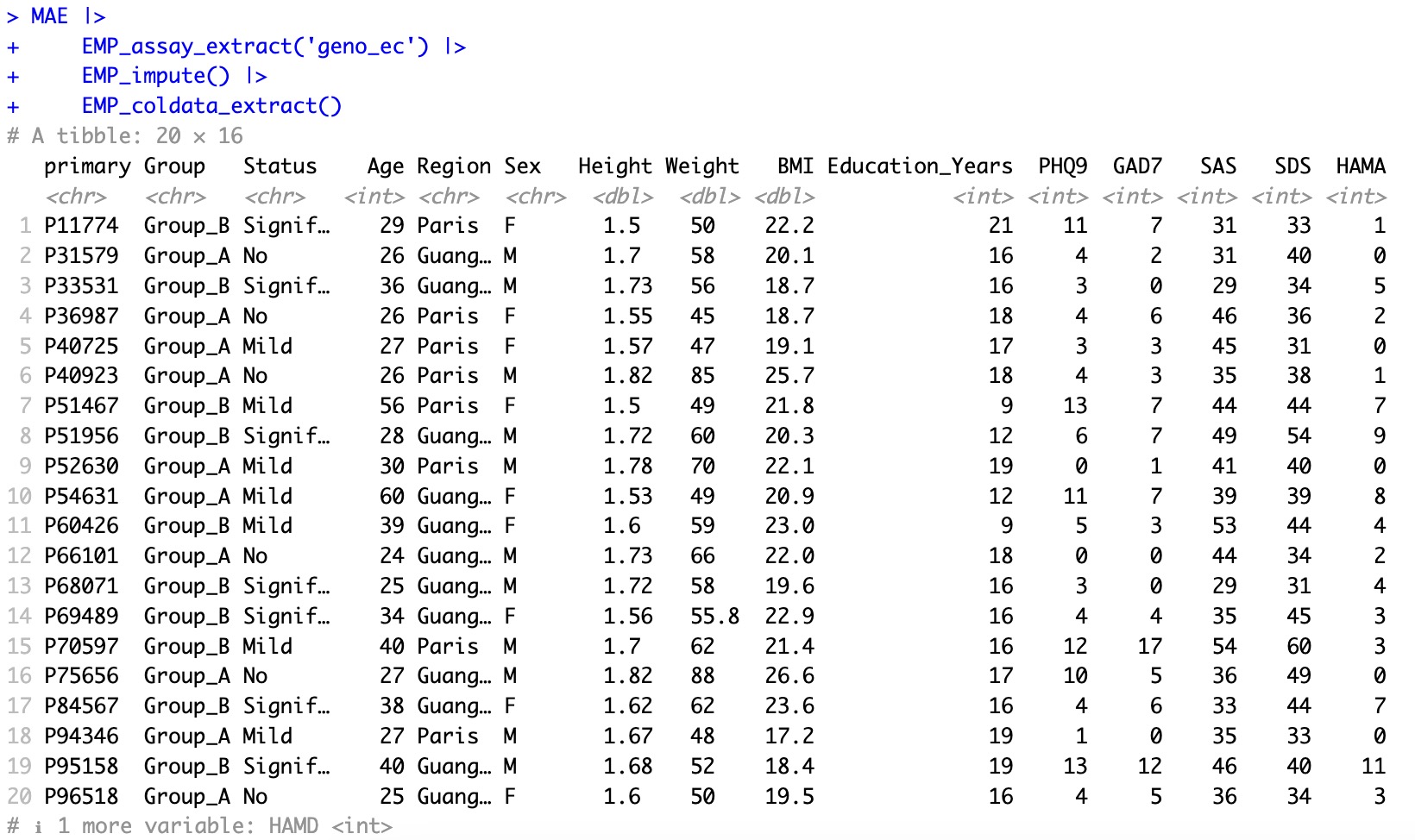
插补后:对coldata的所有缺失值进行插补。
MAE |>
EMP_assay_extract('geno_ec') |>
EMP_impute() |>
EMP_coldata_extract()
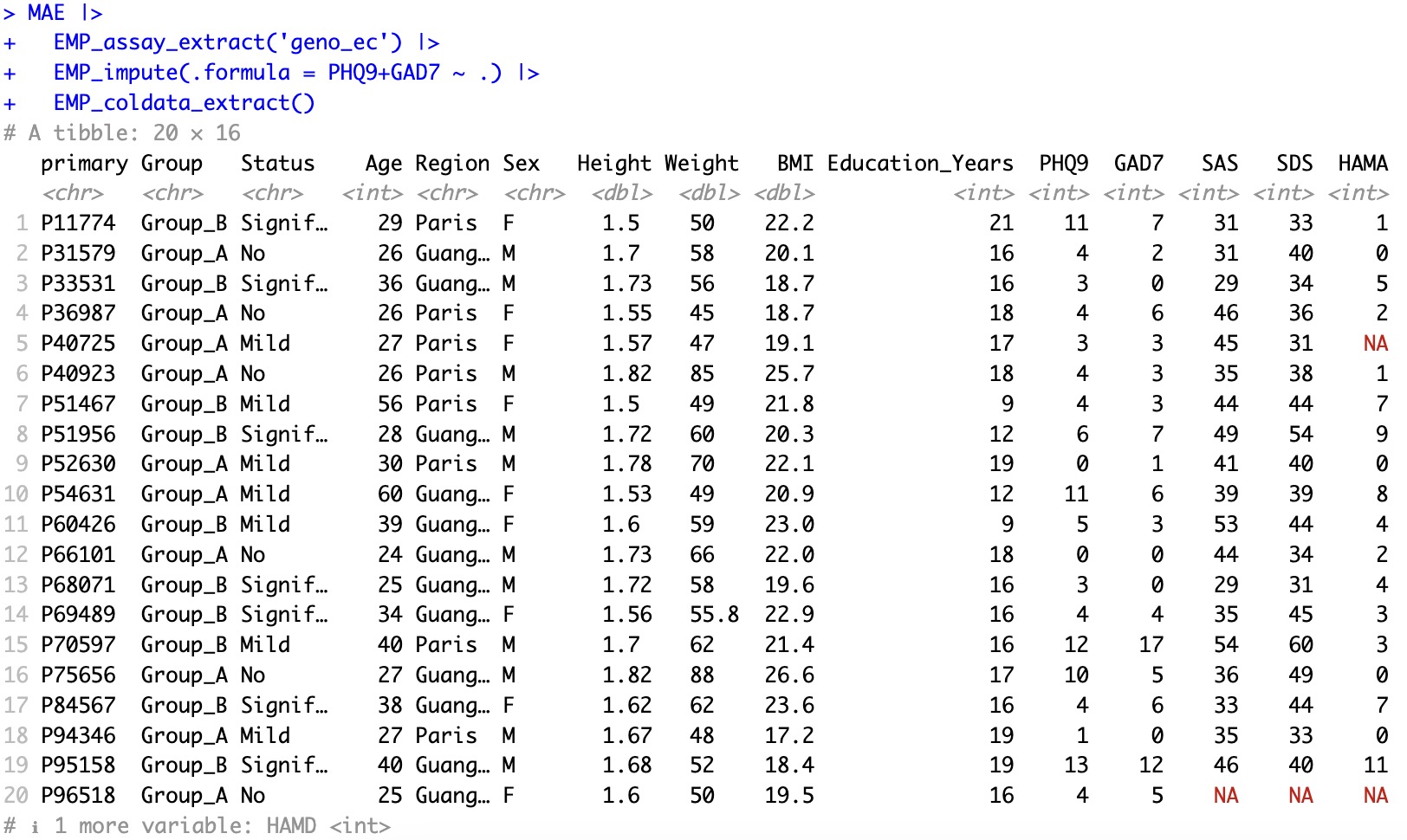
也可以只对coldata的部分缺失值进行插补。例如:只对PHQ9和GAD7的缺失值进行插补。
MAE |>
EMP_assay_extract('geno_ec') |>
EMP_impute(.formula = PHQ9+GAD7 ~ .) |>
EMP_coldata_extract()
5.5.2 插补实验数据assay
当assay不存在缺失值时,使用模块EMP_impute进行插补会提示"Assay data has no NA value!"。
MAE |>
EMP_assay_extract('geno_ec') |>
EMP_impute(coldata = F,assay = T)

5.5.3 插补特征相关数据rowdata
组学项目的rowdata是指特征相关数据,其缺失值的产生主要是由于数据库注释不完善导致。因此,尽管模块EMP_impute支持使用参数rowdata=T进行rowdata的插补,但是插补的结果难以满足实际需要,因此不建议插补rowdata。
5.5.4 插补整个多组学对象的coldata
用户也可以直接对整个多组学数据容器进行表型数据的插补。需要注意的是,此时对象输出的是MultiAssayExperiment。
MAE |>
EMP_impute()
